自己写的fixbatch有效果了,小小激动一下,开心
之前的事情
我周二时候写了个文章,当时是遇到了一个项目问题。是因为批次效应,导致了样本的聚类效果不好,所以我一来是想解决项目问题,二来就想趁此机会,学习下如何矫正批次效应。
当然实际项目并不会给我时间矫正。用svaR包的combat.r矫正后,就已经把问题解决了。
但是最开始的时候,发现了一个问题。就是我明明看到离群的八个样本的表达量箱线图,展现的极值,中位数都和其他正常样本不一样。但是想法也简单,先用中位数矫正的方法矫正下批次效应。看看效果。
preprocessCore矫正效果
但结果就是用经典的R包preprocessCoreR包中位数矫正的结果就是矫正不好。甚至可以说矫正与否没有任何明显变化。只是左右颠倒了下位置,有啥用啊, PCA是相对距离,坐标轴的数字没有太多意义!
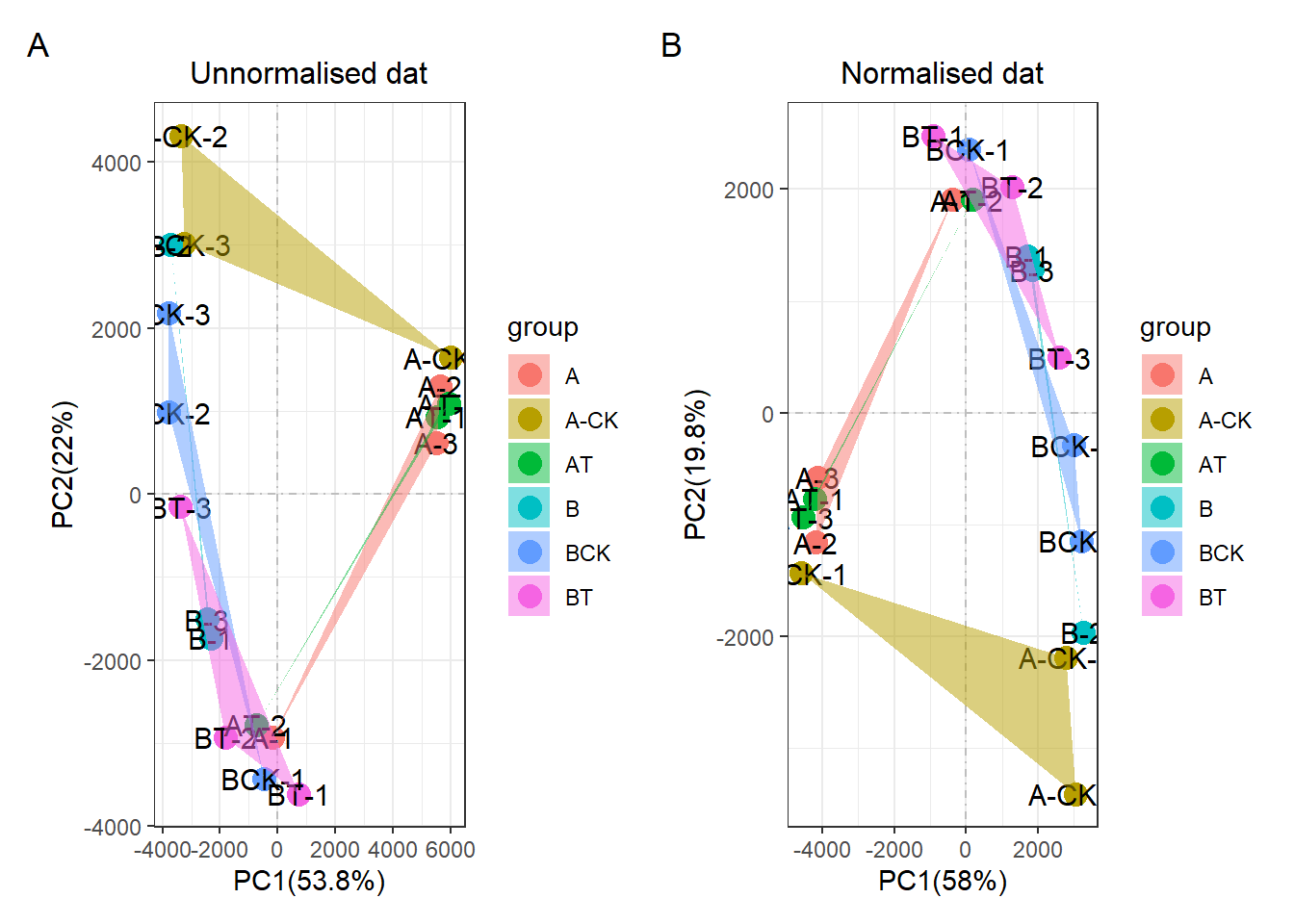
后面我自己不是重新造了一个批次效应的数据,然后用preprocessCore做了矫正,但是效果没有任何变化,如下图,也就是说preprocessCore的中位数矫正的方法可能只是一种类似于标准化一样的功能,只是适用于将缩放到同一个范围尺度内。类似于scale和log一样的功能。
本质上到这里,我就应该放弃用preprocessCore的中位数矫正的思路了。但是我去看edgeR的calcNormFactors函数时候,重新会想起矫正因子的作用,意识到其实edgeR和Deseq2这些差异的R包都会去计算校正因子,从矫正数据分布,然后再做统计检验。
而且他们用的都是,表达量中间,以及差异变化比较小的“稳定基因”来计算,而不是用全部的基因来计算。突然之间,我想到了一个方法,就是用“管家基因”来计算矫正因子。
然后我就分别根据自己之前的一篇文章,利用变异系数以及表达量之和来确定管家基因。并将脚本整理成函数,打包成了R包fixbacth。
实现这个想法花不了多少时间,打包成R包也没画多久时间。但是没想到的时候,自己造的例子可以,如下图。
但是真实的数据。这个利用管家基因的矫正思路是没有任何效果的。PCA矫正前面什么样子,矫正后还是什么样子,最多PC1的百分比变化了几个百分点。
心凉凉的,一直没想到为什么,我决定放弃了。但后面准备写篇公众号,给大家说下,中位数矫正没用的时候。但写着公众号的时候,突然想到:
我算来的的管家基因根本不是管家基因,因为我是把所有样本放在一起计算管家基因,最后得到的肯定是在各个样本中没有任何变化的基因。(因为这个基因只有在离群组和正常组中表达量一致,才会被我选择!!!)
这不是垃圾嘛,我这样算出来肯定是矫正因子在1徘徊啊。肯定矫正没有任何效果,被自己的脑回路打败了~~
所以我需要:
- 区分批次效应分组
- 分别计算每个批次组的“稳定基因”
- 将各个批次组的稳定基因取交集,称为“共有的稳定基因”
- 利用“共有的内参基因”来计算矫正因子
- 再反哺到原始数据上
具体思路如下:
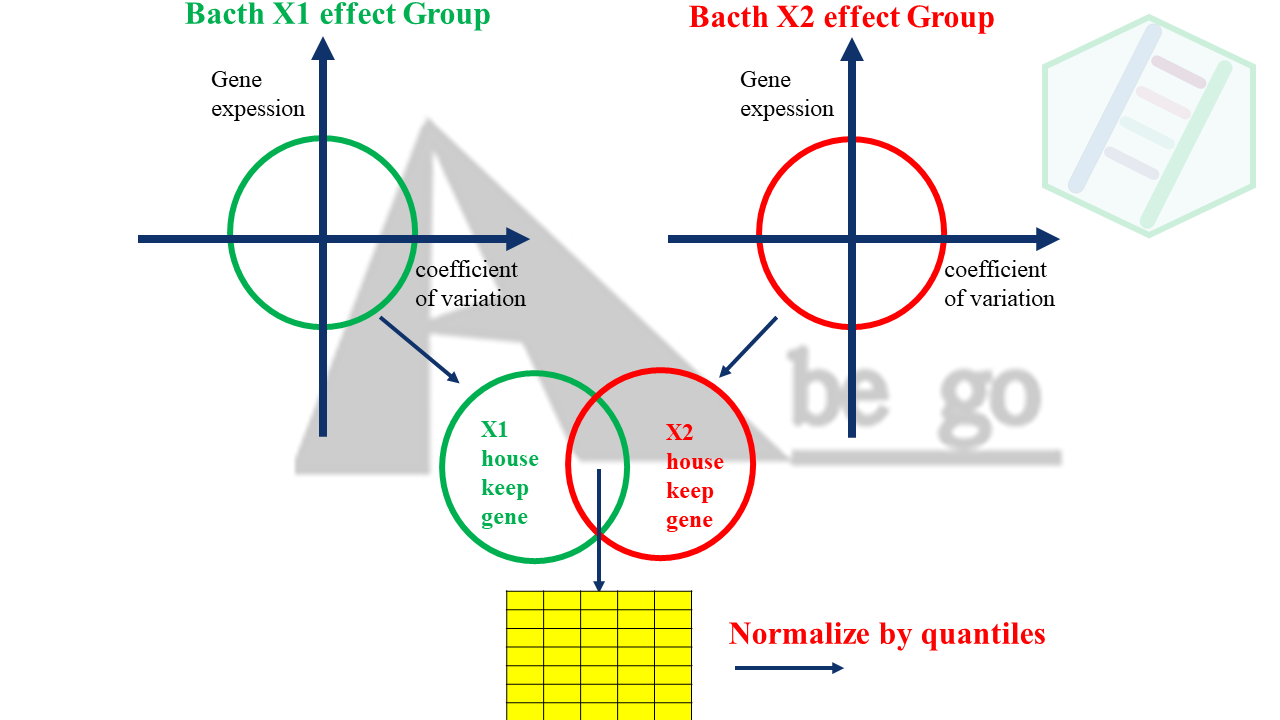
说实话,目的是这样的,用R代码实现起来,困难的一度让我怀疑自己没能力实现自己的想法。虽然我也去Google搜索,但是答案都不让我满意。是,tidyverse我很熟,tidyverse全家桶我肯定实现我的目标。但我不是写脚本呀,我是想做成一个R包。如果我用tidyverse写R包,那我这个R包算是low到不行了,不仅加载一堆附属包,运算速度估计慢的一B。
所以我想用base函数族来实现目标,但是搜到的都是split+apply的解决思路。我觉得太繁琐了,缺乏美感。我就去看sva的源码,发现别人写的代码就是好看。赞叹不已,然后推敲了一段时间,偷学了三行命令。
自己在此基础上完成了构思,目前更新的R包fixbatch已经重新放在GIthub了。
效果非常赞!心满意足,开心到爆炸!
# BiocManager::install("edgeR")
# BiocManager::install("preprocessCore")
# devtools::install_github("wangjiaxuan666/autopca")
# devtools::install_github("wangjiaxuan666/xbox")
# devtools::install_github("wangjiaxuan666/fixbatch")
# install.packages("patchwork")
# set workdir
# 记得更新我的R包奥,要不然以前的BUG,得让你怀疑人生
devtools::update_packages("autopca")
devtools::update_packages("xbox")
devtools::update_packages("fixbatch")
# 加载R包
require(autopca)
require(xbox)
require(fixbatch)
require(tidyverse)
require(preprocessCore)
require(patchwork)
因为我自己构建的数据,离群的是"A-CK-1,A-2,AT-1,AT-3,A-3"样本,所以批次分组如下设计。一定要记得和数据的列名排序对应!
| 样本 | 批次 | 样本 | 批次 |
|---|---|---|---|
| A-1_fpkm | 1 | B-1_fpkm | 1 |
| A-2_fpkm | 2 | B-2_fpkm | 1 |
| A-3_fpkm | 2 | B-3_fpkm | 1 |
| AT-1_fpkm | 2 | BT-1_fpkm | 1 |
| AT-2_fpkm | 1 | BT-2_fpkm | 1 |
| AT-3_fpkm | 2 | BT-3_fpkm | 1 |
| A-CK-1_fpkm | 2 | BCK-1_fpkm | 1 |
| A-CK-2_fpkm | 1 | BCK-2_fpkm | 1 |
| A-CK-3_fpkm | 1 | BCK-3_fpkm | 1 |
下面就是设计批次分组,并开始中位数矫正了
#不矫正的结果
dat_pca_nofix = pca_data_tidy(dat)
p1 <- pca(dat_pca_nofix,rename = "replace",
str_sample = "_fpkm",
str_group = "-\\d$",
display_sample = T,
add_ploy = T) +
ggtitle("Unnormalised dat")
#矫正的结果
bath = c(1L, 2L, 2L, 2L, 1L, 2L, 2L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L)#设置批次分组
dat_df = get_df(dat)#change into data.frame
dat_fix2 = norm_fix(dat_df,bath,expcv = 0.5, exphigh = 0.8, explow = 0.4)
dat_pca_fix = pca_data_tidy(dat_fix2)
p2 <- pca(dat_pca_fix,rename = "replace",
str_sample = "_fpkm",
str_group = "-\\d$",
display_sample = T,
add_ploy = T) +
ggtitle("Normalised dat")
p1+p2+plot_annotation(tag_levels = 'A')
可以看到效果是不错的,开心了,后续我会从GEO数据库里下载一些数据,做下进一步测试,以及利用PC的坐标来做下矫正效果的展示,毕竟现在完全是看图,主观性强。
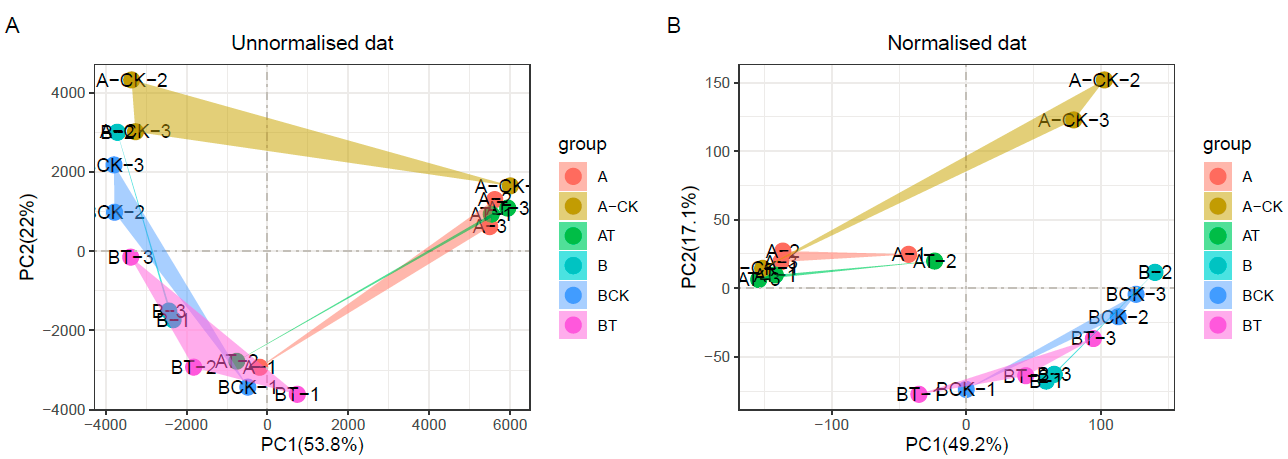
不过现在我也是心满意足了, 毕竟fixbatch这个矫正的脚本算是目前,我最高最水平的脚本之一, 脚本复杂倒是不高, 但好在这个期间, 不断开发自己的想法, 并用一种比较满意的代码(越简单越满意)去实现. 感觉还是非常傲娇的.
具体的代码,大家可以点击原文链接, 到我的Github上查看, 错误之处多谢指点.