从MalaCardsa爬虫收集疾病相关的研究信息📄
MalaCardsa数据库📘,我今天也是第一次知道,感觉这个网站太牛🐃了,收集了各种人类👫疾病的相关信息。基本上把我们手上正在做的项目的背景信息一网打尽。简直就是一直堵车,突然发现捷径的感觉。
近期本身就是在搞空间转录组的项目📊,在做细胞🌕类群注释的时候,遇到的困难还是比较大的。一直都是靠劳动力堆积来做类群注释,耗时费力,但也无可奈何。
尤其我这种本身背景就是搞植物🌴,研究生时候养养小油菜🌱,毕业的工作也就是经常打打嘴炮。基本上对医学的研究背景了解就很少。甚至前段日子看到肿瘤切片和小鼠解剖图片,一度庆幸自己学的是植物…QAQ👴👴。
总之呢,MalaCardsa是个非常牛的网站,强烈安利给医学研究领域的小伙伴。
虽然可能目前很多人没有听说MalaCardsa,但是GeneCards应该或多或少都有用过,或者耳闻过吧。 MalaCardsa就是GeneCards Suite的其中一部分。
GeneCards Suite of Databases包括了:
- GeneCards: A database of human coding RNA (mRNA) genes
- GeneCaRNA: A database of human non-coding RNA (ncRNA) genes
- MalaCards: A database of human maladies and their annotations
- PathCards: The integrated database of human pathways and their annotations
- GeneALaCart: Generates a file of GeneCards annotations for your list of genes
- GeneLoc: Presents an integrated map for each human chromosome, based on data integrated by the GeneLoc algorithm
啧啧啧,这套装有点厉害啊。其中GeneCards和GeneCaRNA提供基因功能的查询,MalaCards提供疾病的背景知识。PathCards研究和绘制基因调控网络,GeneALaCart和GeneLoc提供研究便利,respect!respect!
MalaCards的相关信息💻
MalaCards: The human disease database。MalaCards 是一个人类疾病综合数据库,以GeneCards的基因功能研究,文献调研,以及GeneAnalytics 基因座分析构建的大型疾病数据库。其中包括:症状、药物、物品、基因、临床试验、相关疾病/疾病等等注释信息。
MalaCards疾病数据库集成了21787个疾病条目,包括罕见疾病、遗传疾病、复杂疾病等。
我们以肝病为例,进入MalaCards(https://www.malacards.org/#)后,在搜索框输入“Liver Disease”,然后进入如下页面.
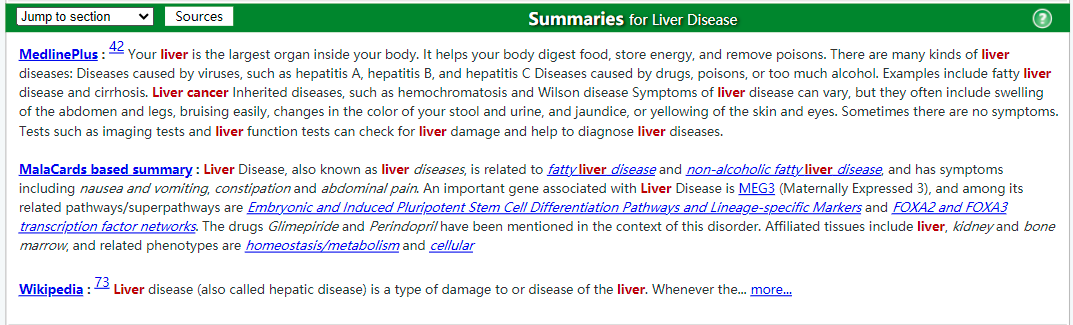
接着往下展示的是肝病相关的基因,并附带了一个网络图可视化,但实话说,图很丑。
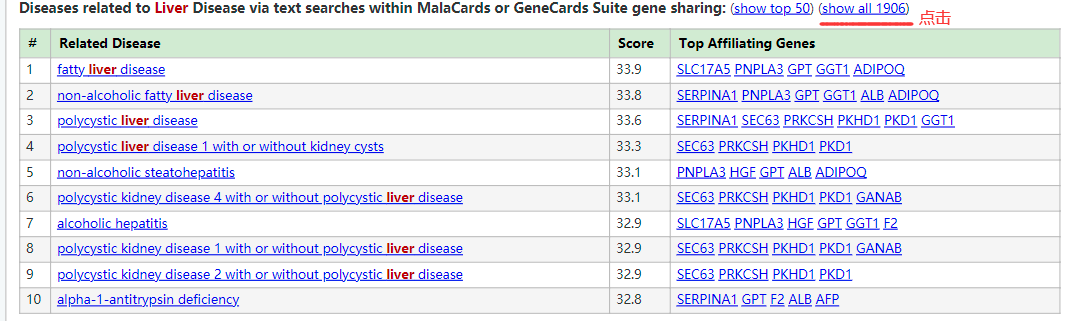
以及还有肝病相关的临床症状,这个对背景知识应该是非常及时的补充。但是如果想看全部结果,需要点击show all。这个选项对后续的爬虫分析也很重要,所以如果你想爬虫获得最全的信息,最好点击下show all。
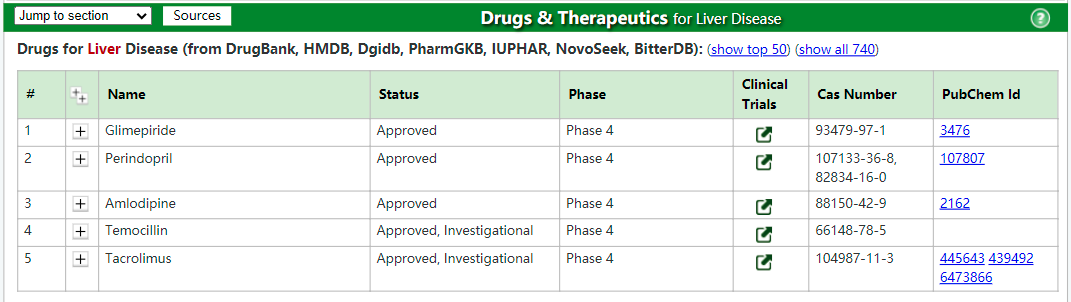
另外最想不到的是,MalaCards还收录了疾病的治疗药物,这个信息如果利用的好,估计能为文章填色不少。
用python收集MalaCards的信息🐛
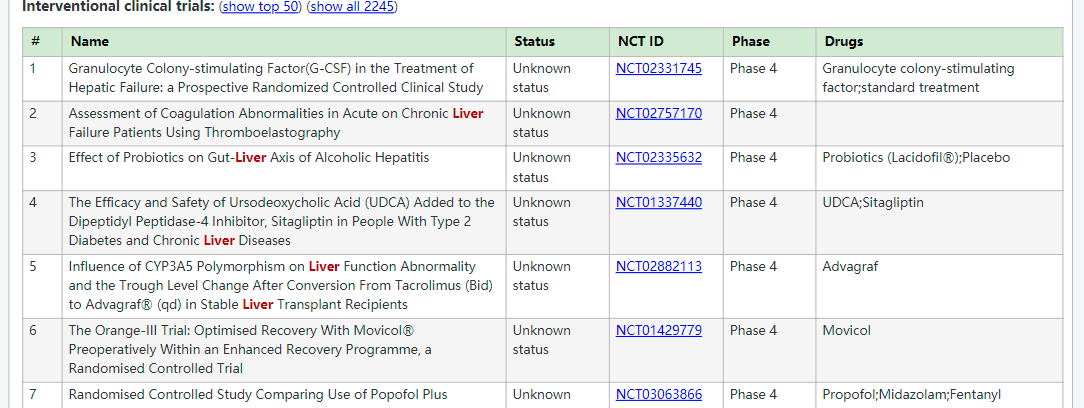
当然,MalaCards数据库还整理了和肝病相关的研究文章目录,这个就不推荐show all,因为文章太多了。所以就不再推荐了。除了收集文章以外,数据还从文章中收集了肝病相关的基因列表。
这个简直太重要了,我们就是想找到一些marker基因来做细胞类型的注释,另外找到一些marker基因去讨论功能和疾病发展。
如图所示,但是一定要记得点击show all,这个对后续爬虫也很重要,否则就会遗漏信息。
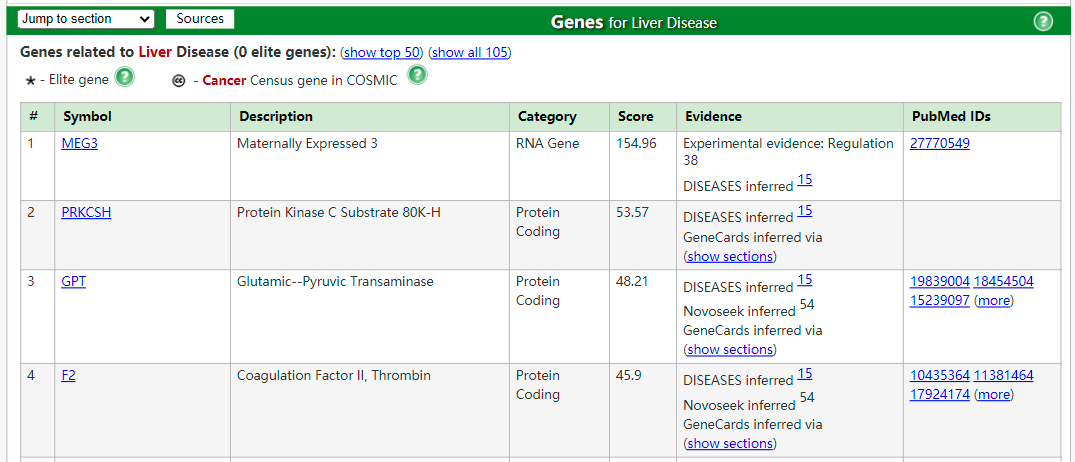
那么除了疾病相关的基因以外,还收录了和疾病相关的结构变异(SNV)以及拷贝数变异(CNV),简直了,只能说一句:“”强强强“。
当然GO,KEGG的注释分析当然也不会少。可见MalaCards是非常值得爬虫(贱兮兮笑下)。
但其实我之前写过爬虫🐛准备爬GeneCards的功能注释信息,但是很不幸。GeneCards应该有反爬虫机制,所以爬虫时而成功时而失败。关键是当时爬取的是基因的功能注释,也就是说每查找一次基因就需要刷新窗口一次,几百个基因,这种动不动就报错的脚本,这谁顶得住,遂放弃。
但是这次只是需要一个网页就可以收集所有信息,为了应对发爬虫机制。选择直接在浏览器上点击,ctrl+S下载,就是这么流畅。
Python脚本放在GIthub上,微信公众号需要点击阅读原文可直达Github。下载好Python脚本后,将脚本移到工作目录,建议malacards的本地网页也放在同一目录下。
强烈安利Vscode 宇宙第一代码编辑器🐍。
# 需要把get_info_from_malacard.py放到工作目录下
import get_info_from_malacard as gf
# 读取本地下载的html文件
html = "~/xxxx/xxxx/malacards.html"
soup = gf.read_file(html)
# id是想要调取的信息,我已经整理好了
id = ["RelatedDiseases-table",
"MaladiesUnifiedCompounds-table",
"ClinicalTrial-table",
"Publications-table",
"RelatedGenes-table",
"ClinVarVariations-table",
"CnvdVariations-table",
"de_genes-table",
"Pathway-table",
"go_proc-table"
]
for id in id:
gf.get_info(soup,id)
# 自动生成结果文件,保存在工作目录下的excel文件中
今天就到这里了,完结~,欢迎交流!