脚本的目的
大家首先会觉得为什么要写这种脚本。提起来就没什么用,基因组注释文件不是从NCBI-Genome或者Ensembl这些基因组网站上直接点击链接就可以了吗。
其实这话也不全对, 如果想去下载一些病毒和细菌这类小基因组文件的时候,往往只能在NCBI的Nucleotide数据库找到。然后就会发现网页是这样子的:
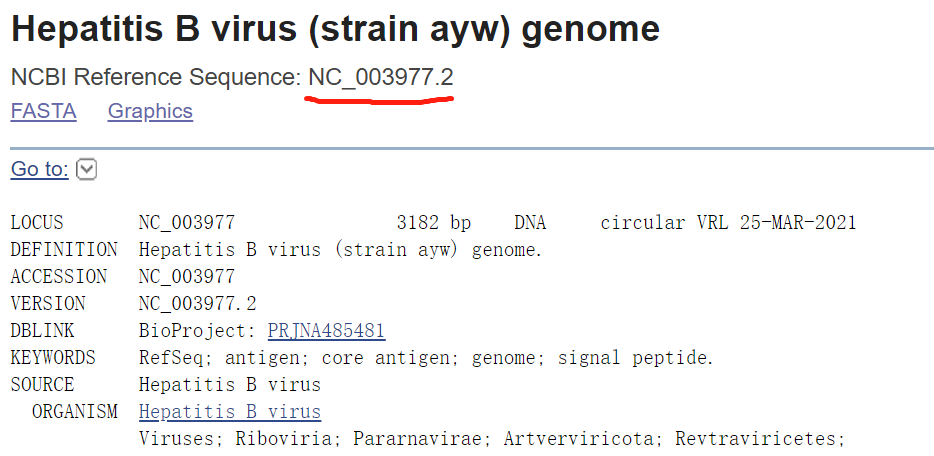
简而言之,就是虽然fa文件可以网页上点击下载,但是gtf文件却找不到地方下载,所有的注释信息都是在网页上以很冗余的形式表示出来。这种情况针对特定研究领域的初学者(嗯,没错就是我自己)还是相当困扰的。我觉得这个情况肯定是很普遍的,不会只有我一个人遇到这样的问题。
就拿我上份工作🏢🏢🏢来说,这种情况我都遇见过了两三次,只不过当时直接提交给分析人员。自己不做所以不操这份心。 不当家不知菜米油盐贵
但我肯定不会费大力气去写爬虫🐛🐛🐛调取的,毕竟我觉得常用的功能,应该有人写过脚本,拿来全然不费功夫。然后我相当有信心的去谷歌了。很奇怪,搜中文,没结果…。基本上没见有人提到这个问题(难道没人遇到这个问题还是说另有捷径❓❓❓)。最后搜英文,总算最终找到了解决方法,其实很简单就是一个链接输入调参而言。
虽然结果很简单,但是为了这个问题搜索的过程却不是那么轻轻愉悦的,做个分享利己利人💾💾💾,如果有需要的朋友,可以去我的Github上下载down_genome_from_nucleotide.sh,链接点此。
脚本的使用
因为是纯shell脚本📗📗📗,可以直接用在Linux系统上运行,用Windows的同学需要在Win10下面安装WSL,也就是Win10的linux子系统,效果是一样的。
# wget "https://raw.githubusercontent.com/wangjiaxuan666/WeChat-pubword/master/down_genome_from_nucleotide.sh"
# 下载好后,当面目录下会出现down_genome_from_nucleotide.sh文件
# run
bash down_genome_from_nucletide -i NC_003977.2 # NC_003977.2就是需要输入ID,见上图的红线处标识
# ok
一步搞定!运行完成后,会在屏幕上显示:
==================
The genome has download in NC_003977.2 files !
==================
这个时候,检查当前目录就会多了一个NC_003977.2的文件夹🐍,其中就会有我们需要download的基因组序列文件.fa和基因组注释文件.gtf。
切记参数是-i,如果输入其他参数不会识别,并且会报错:
==================
please check the '-i name' again
==================
注意name是NCBI Reference Sequence的编号,可以在Nucleotide的结果网页中查到。
今天就到此结束了,撒花!