第二个R包!
密度热点图
这个事情起因是客户想要一个能够展现单细胞数据和普通转录组数据之间相似性的图。我推荐客户,可以考虑画一个散点图, 横坐标是单细胞数据,纵坐标是普通转录组数据。再用相关性来做检验相似性强弱。
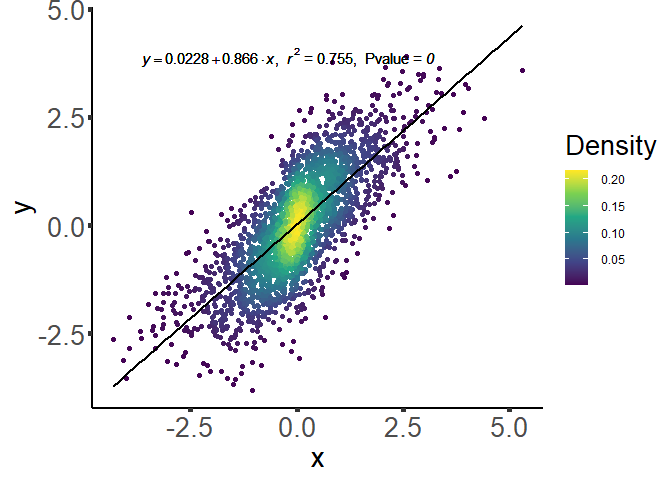
最开始是没想画密度热点图的,但是因为后面又看到一个文章。觉得自己想尝试一下,就用ggplot2包的geom_hex和stat包 的cor.text以及lm进行了尝试了。结果还可以!下图就是我自己造的数据生成的例图。大概就是这个样子的。只是细节可能还会调整一些。
但是 geom_hex并不是我想象中最优解,只是当时客户催的紧要结果,而且实话说我也没有太多时间去摸索。就交付了。觉得不是最优解的原因是
- geom_hex统计每一个六边形区域内的点数,并不是每一个数值都有一个点在图中。简单理解,就是你有1000个数据画图,geom_hex可能就给你10个六边形(这是完全可能的),数据多的时候不明显,数据一旦少了就会非常明显。
- 颜色变化不显著,因为是算一个hex中的频数,所以差异不会很明显。
但是我们还是可以知道,密度热点图还是非常好看的,不是吗,遇到展示两组数据相似性的时候,帮助是非常大的。
自己造R包
五一时间,第一天去了新房子里钻孔,拧水龙头(自我感觉越来越熟练)。第二天去了上下九溜达了。第三天想着还是把这个脚本再完善一下。所以就鼓捣了一个R包。这个R包本质上不是为了画密度热点图。而是为了收录自己平时写的一些实用小脚本的。
R包,我取名为xbox,目前收录了做数据框的列之间的T检验的``col_ttest,以及转置数据框,让行列互相颠倒的df_t, 还有根据KO id,给出KEGG数据库三个层级注释的koid_to_pathway,其次还有tibble和data.frame之间转换的get_tb和get_df`。具体的用处和例子可以看帮助文档或者Github上的说明。点击阅读原文可以跳转Github。
今天新增的功能就是画密度热点图的heatpoint和plot
例子如下:
points = 5000
x = c(rnorm(points/2))
y = x + rnorm(points/2,sd=0.8)
x = sign(x)*abs(x)^1.3
heatpoint(x,y) -> dat_result
str(dat_result)
#> List of 3
#> $ plot.data :'data.frame': 2500 obs. of 3 variables:
#> $ cor.result:List of 3
#> $ lm.result :List of 3
#including the three list the plot data
head(dat_result$plot.data)
#> x y Density
#> 1 -0.115142560 -0.08891457 0.20889658
#> 2 0.001437608 -1.24490133 0.08753891
#> 3 -0.229579517 -0.90045187 0.14522626
#> 4 1.381172256 1.48550963 0.05487725
#> 5 1.114704663 2.09831530 0.03998420
#> 6 -0.910906938 -0.25959069 0.07883229
# and the result of cor
data.frame(dat_result$cor.result)
#> cor pvalue method
#> 1 0.7552128 0 Pearson's product-moment correlation
# and the result of lm
str(dat_result$lm.result)
#> List of 3
#> $ formula: Named num [1:2] 0.0228 0.866
#> $ method : symbol lm
#> $ exp :Class 'formula' language y ~ x
可以看到,heatpoint给出了相关性检验的数据以及线性回归的表达式以及画图数据,如果这个时候,你对R语言中的画图已经有了一定基础,完全可以自己用ggplot2或者base来进行绘图。简单点的话,可以直接用我做好的plot进行绘图,一键出图!
plot(dat_result)
当然,自动化也有自动化的傻点,检验数值是根据画图的长宽来自动调整的,改变图片宽度,既可以规避表达式和点重合。
Heatpoint参数
plot没设置什么参数,但是heatpoint设置了很多参数了。只是大部分参数都设置了默认值。其实heatpoint(x,y) -> dat_result等价于:
dat_result <- heatpoint(x,y,xlim = NULL,ylim = NULL,log = "",grid = 100,only = "none",method = "pearson",formula = y ~ x,...)
其中xlim和ylim是设置计算的范围,原因在于并不是所有输入的数据都希望被计算密度。而log是是否需要对数据进行log10标准化。这个对组学数据是很常用的,毕竟大部分数据都是很小的,只有极少数基因是高表达的。如果想尽可能把低表达基因和高表达基因放在一起展示。那么log10标准化是必不可少的。grid就是设置分区的参数,简单来讲就是把输入数据分成几个部分,分别计算密度。一个区域内的点共享密度值。method和formula就分别是相关性的方法和线性回归的表达式了。
附
最开始没想造R包的,本质上是当个调包侠的。但是搜来搜去都没有什么好答案。最贴近的答案是网上一个叫Kamil Slowikowski的教程。链接是https://slowkow.com/notes/ggplot2-color-by-density/.
BUT 我好像没测试成功在我的数据上,也就是说你跑他提供的例子是没问题的,但是我自己的数据就不行了。所以继续寻找。最后找到了一个非常牛的R包-LSD。讲实话,我看这个LSD的源代码看了挺久的,我觉得这个作者真的是很牛。以后继续学习吧。
回到正题上,LSD提供了一个可行的方法,叫做heatscatter,但是这个函数是只出图,并且是base::point画图。这样不仅我会嫌弃它画图丑,还嫌弃我自己不会base画图,所以我也改不了,没办法让结果按照我的想法改进。
所以,仔细学习了作者的代码,然后做改进,加入线性回归和相关性检验. 将计算和画图功能分开,用ggplot2进行可视化。完美!
目前heatpoint功能已经整合在xbox包里了,放在gitub上,欢迎使用并提出宝贵意见。