其中, 启动子凝聚物是在细胞核中的自发产生的瞬时动态结构,其形成主要依靠转录因子的浓度。相分离的浓缩假说解释了不同的转录因子如何能够招募相同的Pol II聚合酶。启动子凝聚物是高度动态的,并且可以在细胞核内, 发生位点改变,收缩和重整。
基因转录组调控途径(译文)

作者Patrick Cramer简介:
- 《cell》的编辑,EMBL的成员
- 哥廷根马克斯普朗克生物物理化学研究所所长
- 确定了RNA聚合酶II的三维结构,这项工作也直接促进他的博后老板因为研究真核转录调控获得06年诺贝尔奖。
- 欧洲科学院院士,德国国家科学院院士
背景
受调节的基因转录决定细胞的种类和功能。最近的分子结构研究已经证明了在起始和延伸阶段,
通过RNA聚合酶进行转录调控。科学院通过显微镜观察到,
作者认为这种转录和调控的模式可以在很长一段时间内作为科学家的[共识](# section01)
这个condensates咋翻译啊, 可真是惆怅
在生物体的发育过程中,特定基因在不同的细胞中表达,以建立不同类型的细胞。这需要基因表达的复杂调节. 因此,了解基因调控需要详细了解转录机制。
在过去的几十年中,分子和细胞研究已经阐明了进行转录因子的结构,并提供了各种关于如何调节这些转录复合物的见解和假说。
目前共识是: 转录是通过RNA聚合酶进行的,依赖DNA模板进行RNA的产生。为了启动转录,RNA聚合酶需要识别基因的启动子区域。然后在拓扑酶的作用下打开DNA双链体,开始合成RNA并从启动子中逃逸。并且得到的延伸复合物进行延伸RNA链. 直至其达到终止信号并释放DNA和RNA。
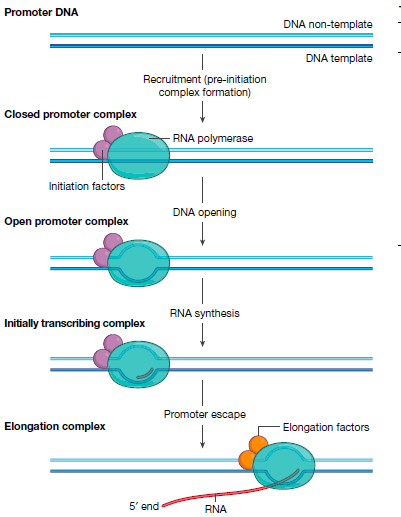
半个世纪前,从真核细胞中分离出3种RNA聚合酶1 。后来发现这些聚合酶可以转录不同类别的基因。RNA聚合酶Pol I产生大的核糖体rRNA前体,Pol II合成信使RNA和多种非编码RNA,Pol III产生转移RNA和小核糖体RNA。聚合酶的相关因子和调节机制不同。
植物还有聚合酶IV, 和聚合V, 与sRNA介导基因的甲基化沉默相关
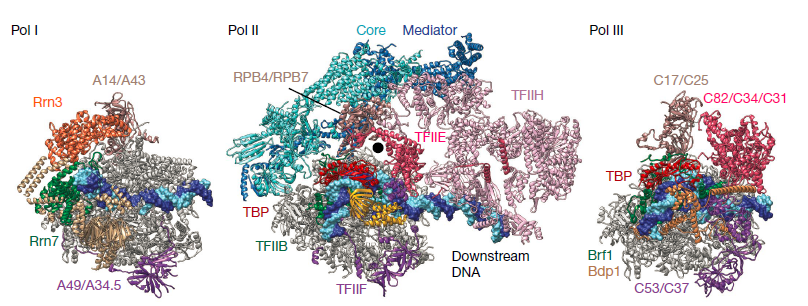
首先看一下, 三种聚合酶的形态.核糖体上还会结合不同的因子来调控和发挥作用.
为了启动转录, 聚合酶首要第一点就是被招募到在基因的转录起始区域, 但是核小体会抑制转录的发生, 其原因也是在这里. 只有当核小体转移或者移除的时候, 转录才会发生[[1-3]](# section02). 目前研究也表型在核小体缺失的区域更容易发现活性的转录因子.
人类的启动子区域一般有CpG岛, 这些CpG岛回抑制核小体组装和结合, 从而促进聚合酶的结合. 但是这样的启动子特征主要是针对管家基因. 一般情况下, 特定细胞类型里只有一小部分的POL ii 启动子有活性, 这些启动子区域是被转录因子以序列特异性的识别而激活 . 从而指导聚合酶到特定的DNA元件上进行转录. 转录因子是一类具有低复杂程度的氨基酸序列. 能够招募聚合酶, 调控启动子可及性的蛋白质. 人类已知的转录因子大约有1600种, 大部分转录因子可以和裸漏的DNA结合, 但是一小部分是可以与封闭的核小体DNA结合. 后者往往是一类特殊的, 起到前锋作用的转录因子. 率先在局部开放染色体以实现转录.
这一步通常涉及到组蛋白乙酰转移酶和染色质重塑复合物的募集
转录因子除了可以结合启动子以外, 还可以结合增强子. 增强子是一类远端调控元件, 距离调控转录目标区域非常遥远的一类DNA元件. 增强子甚至可以离靶基因超过1M以上碱基的距离.
增强子区域往往也会包含多个协同转录因子的结合位点. 因此某些活性增强子还能够产生增强子RNA, 而且有意思的是, 这种转录是可以发生在前后两个方向上的. 对于转录因子, 有的时候也能观察到这种双向转录.
这就意味着, 转录因子结合位点不是一定就调控DNA序列下游的靶基因, 很可能是对DNA序列上游的靶基因进行调控的.
增强子往往只会存在基因组的特定区域, 作者认为这种机制是与染色体的空间结构相关的, 并成为拓扑结构相关结构域.
细说RNA聚合酶
最简单的RNA聚合酶是来自噬菌体的, 只有一个单一的多肽. 但是仍然需要两个额外的起始转录因子, 以及和转录延伸因子才能发挥转录功能. 其他细胞的RNA聚合酶也需要其他因子. 并且聚合酶本身还是多亚基的复合体. Pol I,Pol II和Pol III分别含有14,1,12和17个亚基。两个最大的子单元以三种银色和金色显示. 较小的亚基以各种其他颜色显示。如聚合酶II结构所揭示的那样,两个大亚基形成保持活性中心一个裂口,并且较小的亚基围绕外周排列.
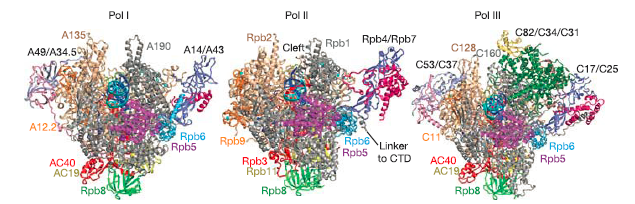
聚合酶的关键元素是夹子(可移动并且可以关闭活动中心),以及结合TFIIB及其相关起始因子的壁和坞。而植物细胞含有另外两种Pol-II样聚合酶(Pol IV和Pol V),它们参与基因沉默.
转录的过程
转录起始
聚合酶只有三种(动物), 本身他们是不会识别启动子的, 更别说启动子本身还是相对多变的DNA序列元件. 所以聚合酶需要转录起始因子来搭建桥梁. 聚合酶和转录起始因子聚在一起形成特异性起始复合物(PICS). 然后转录起始因子结合在转录起始位点上游的DNA序列上, 而聚合酶的活性中心-裂缝则定位在下游DNA中.
DNA的解螺旋
聚合酶II是需要额外的酶-DNA拓扑酶XPB进行DNA的解螺旋. 但是聚合酶I和聚合酶III却可以诱发DNA自发性解螺旋.作者推测原因是由于在聚合酶I和III的解螺旋过程中是有大量的蛋白参与的, 对DNA起到了很好的保护作用. 但是聚合酶是发挥mRNA转录的重要功能, 其缺乏这些大量蛋白参与, 就丧失了稳定DNA的元素, 所以只要额外的解螺旋蛋白参与其中, 并稳定DNA的结构.
转录的发令官
转录的三种途径都会受到调控和监管, 其中设计到转录起始因子的修饰化转变, 比如很多转录相关的蛋白需要发生磷酸化才能发挥功能等. 而这些能够带来磷酸化基团的激酶就是起到了转录调控的发令官的作用.
延伸的调节
转录复合物以渐进的方式转录RNA链。转录组复合物通常包含位于DNA气泡内的DNA-RNA杂交双链体. 当聚合酶到达一个需要添加核苷酸的位置, 聚合酶封闭活性位点, 通过金属离子催化位点催化磷酸二酯键的合成. 并且转移到下一个位点.
这个过程并不总是顺顺利利的, 反而是一波三折, 有很多因素都可以调控转录复合物的延伸
- 特定的__DNA序列__可以阻滞转录复合物的前进
- __核小体__会阻遏POL II转录复合物的前进, 但是转录延伸因子TFIIS可以再次启动转录. POL I和POL III 也有这样的延伸因子可以挽救这种阻遏.
- 后生动物(除了原生动物以外的动物)甚至可以发生转录的__延伸相位__变化(啥意思?转录跳跃?)
- 而且研究表明聚合酶POL II 特别容易在转录起始位点下游50bp处暂停, 而且这个机制是被广泛调控的.
- 值得注意的是, 转录延伸的这个过程中是设计到空间构象的改变(这不废话嘛), 所以还收到构象的调控
所有因子的聚集
由于需要大量因素,因此出现了如何在细胞核中组织转录的问题 - 即如何快速递送所需因子,以及如何将起始和延伸因子分开。
传统的认为是转录发生在特定的区域内, 例如人们很早就知道转录发生在无核膜隔室(核仁)区, 也在显微镜下观察下, 提出Pol II转录过程发生在被称为核枢纽或者转录工厂 的区域内。活细胞超分辨率显微镜随后揭示了Pol II的动态病灶成像,也再次验证了这一点.
但目前又有新的假说, 认为转录过程的因子聚焦在一起是因为细胞内液相的原因, 简称相分离.原理我也不懂就不再过多说了. Skip, Skip
转录活动的模型 {# section01}
综上所述, 科学家提出了Pol II转录活动的简化假设模型。下图所示, 在该模型中,动态启动子凝聚物含有转录因子,共激活因子,未磷酸化的Pol II酶和起始因子。转录基因区凝聚物还包括染色质重塑和修饰的因素。黑色箭头表示转录起始位点(TSS),黑色框表示多腺苷酸化(pA)位点。Pol II从左(上游)移动到右(下游)。
注意,为清楚起见,省略了核小体 - 染色质复合物:
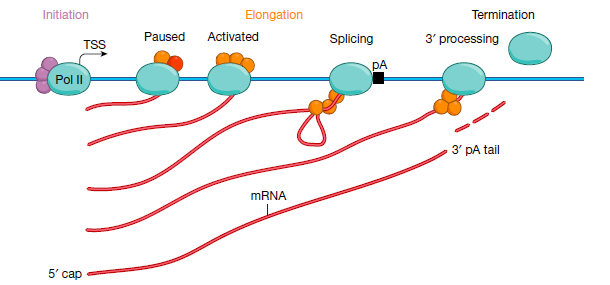
这里面有两个很重要的概念, 一个是动态启动子凝聚物, 一个是转录基因区凝聚物. 这是因为目前大部分科学接受的理论是, 转录通过生物大分子的无序结构形成相, 相与相之间互作促进转录所需因子的聚集. 由此形成不同功能通途的凝集物:
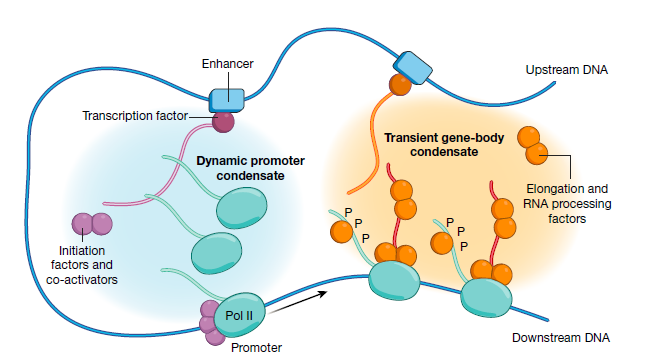
其中浅蓝色的就是
动态起始凝聚物, 浅黄色的就是转录基因区凝聚物.
其中, 启动子凝聚物是在细胞核中的自发产生的瞬时动态结构,其形成主要依靠转录因子的浓度。相分离的浓缩假说解释了不同的转录因子如何能够招募相同的Pol II聚合酶。启动子凝聚物是高度动态的,并且可以在细胞核内, 发生位点改变,收缩和重整。
基因体凝聚物通过转录聚合酶来形成, 发生在启动子序列下游区域。基因体凝聚物的浓缩受到磷酸化,甲基化,乙酰化等蛋白修饰的调控,原因是这些可变的修饰可以影响参与到相分离的蛋白活性和状态.
不同的凝聚物之间可能共用一部分蛋白, 这也能解释为什么会存在部分转录因子和增强子可以调控两端的基因转录. 随着研究的不断深入, 后面还可能会发现新的凝聚物的存在.
我们在回过来看上图的转录模型, 首先是转录因子结合在启动子或者增强子区域, 招募启动子凝聚物, 启动子凝聚物中有转录起始所需的转录起始因子和未发生磷酸化的聚合酶. 随后在激酶的催化下, 聚合酶被磷酸化, 招募转录基因体凝聚物. 其中包含了延伸因子和拓扑酶等转录延伸所需要的因子. 其中磷酸化的聚合酶依靠DNA模板, 在凝集物中穿梭编码RNA.
完结撒花. 这篇文章原文还涉及一部分关于转录过程所需因子的列表, 因为我不需要了解那么细致, 就没收集, 如果做这方面的, 比较感兴趣的可以看原文
附:(常用的转录抑制药物以及原理)
参考文献 {# section02}
- Lorch, Y. & Kornberg, R. D. Chromatin-remodeling for transcription. Q. Rev.Biophys. 50, e5 (2017).
- Knezetic, J. A. & Luse, D. S. The presence of nucleosomes on a DNA template prevents initiation by RNA polymerase II in vitro. Cell 45, 95–104 (1986).
- Lorch, Y., LaPointe, J. W. & Kornberg, R. D. Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones. Cell 49, 203–210 (1987).